Summary
Marc J Williams
2024-01-05
- 1 Filtering
- 2 QC
- 3 Summary
- 4 Summary per cell type
- 5 Copy number landscape
- 5.1 Full cohort
- 5.2 BRCA2 hets
- 5.3 BRCA1 hets
- 5.4 WT
- 5.5 Per patient
- 5.6 Per patient (individual plots)
- 5.6.1 ATMHET6182
- 5.6.2 B1HET6139
- 5.6.3 B1HET6301
- 5.6.4 B1HET6389
- 5.6.5 B1HET6550
- 5.6.6 WT6536
- 5.6.7 B2HET16
- 5.6.8 B2HET18
- 5.6.9 B2HET21
- 5.6.10 B2HET23
- 5.6.11 B2HET25
- 5.6.12 B1HET43
- 5.6.13 B1HET49
- 5.6.14 WT12
- 5.6.15 WT28
- 5.6.16 WT6
- 5.6.17 WT7
- 5.6.18 WT8
- 5.6.19 WT6520
- 5.6.20 B1HET6410
- 5.6.21 B1HET6537
- 5.6.26 B1HET17
- 5.6.27 WT6752
- 5.6.29 WT62
- 5.6.31 B1HET6548
- 5.6.32 B2HET6532
- 5.6.34 B2HET232
1 Filtering
For the analysis shown here cells that fulfill the following criteria have been included:
- Quality score > 0.75
- Number of reads > 100,000
- Not in s-phase (from s-phase classifier)
- Cell has diploid baseline ploidy (some high ploidy cells may be replicating cells not caught by the s-phase classifier)
A cell is classed as aneuploid if it has 1 or more chromosome arm that is non-diploid. This therefore classifies cells that may have small focal gains or losses as “normal”. Some cells appear to have quite noisy profiles so requiring a whole chromosome arm to be aberrent mitigates noisy cells inflating the number of aneuploid cells.
There are a number of what seem to be genuine high ploidy cells but many have likely incorrect ploidy calls. So for now we’ll just remove any cell that has baseline ploidy != 2. We’ll need to return to this and come up with a strategy to ensure we include genuine high ploidy cells.
After filtering as described above these are the number of cells per sample:
| sample | sphase_FALSE | sphase_TRUE | total |
|---|---|---|---|
| ATMHET6182 | 760 | 249 | 1009 |
| B1HET17 | 591 | 2 | 593 |
| B1HET43 | 4224 | 522 | 4746 |
| B1HET49 | 3576 | 721 | 4297 |
| B1HET6139 | 133 | 1 | 134 |
| B1HET6301 | 171 | 35 | 206 |
| B1HET6389 | 469 | 0 | 469 |
| B1HET6410 | 1009 | 6 | 1015 |
| B1HET6537 | 1476 | 2 | 1478 |
| B1HET6548 | 1068 | 7 | 1075 |
| B1HET6550 | 1745 | 1 | 1746 |
| B2HET16 | 2556 | 679 | 3235 |
| B2HET18 | 1815 | 490 | 2305 |
| B2HET21 | 2985 | 857 | 3842 |
| B2HET23 | 4663 | 1308 | 5971 |
| B2HET232 | 666 | 4 | 670 |
| B2HET25 | 2002 | 939 | 2941 |
| B2HET6532 | 1208 | 1 | 1209 |
| WT12 | 170 | 49 | 219 |
| WT28 | 201 | 37 | 238 |
| WT6 | 2625 | 736 | 3361 |
| WT62 | 2010 | 17 | 2027 |
| WT6520 | 1071 | 3 | 1074 |
| WT6536 | 884 | 0 | 884 |
| WT6752 | 1213 | 0 | 1213 |
| WT7 | 845 | 165 | 1010 |
| WT8 | 172 | 10 | 182 |
And by sample type
| sampletype | sphase_FALSE | sphase_TRUE | total |
|---|---|---|---|
| ATM-Het | 760 | 249 | 1009 |
| BRCA1-Het | 16370 | 1346 | 17716 |
| BRCA2-Het | 19347 | 4309 | 23656 |
| WT | 11698 | 1037 | 12735 |
And per sample per cell type:
| sample | LUMBAS | VFTISS | BAS | LUM | BL | LUM-B2 | AV | HS | BA | BAS-LEFT | BAS-RIGHT | LUM-LEFT | LUM-RIGHT | MIX | LUM-M | LUM-P | TISS |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ATMHET6182 | 228 | 781 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET17 | NA | NA | 574 | 19 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET43 | NA | NA | 938 | 1282 | 1369 | 1157 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET49 | NA | NA | 1451 | 488 | 1037 | 1321 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6139 | NA | 134 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6301 | NA | 203 | 1 | 2 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6389 | NA | NA | NA | NA | NA | NA | 469 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6410 | NA | NA | 333 | NA | NA | NA | 380 | 302 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6537 | NA | NA | NA | NA | NA | NA | 682 | 796 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6548 | NA | NA | NA | 1075 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6550 | NA | NA | NA | NA | NA | NA | 629 | 890 | 227 | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET16 | NA | NA | 353 | 1088 | 914 | 875 | NA | NA | NA | NA | NA | NA | NA | 5 | NA | NA | NA |
| B2HET18 | NA | NA | 1383 | 471 | 89 | 305 | NA | NA | NA | NA | NA | NA | NA | 57 | NA | NA | NA |
| B2HET21 | NA | NA | 1948 | NA | 1892 | 2 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET23 | NA | NA | 1294 | 1030 | 2181 | 1466 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET232 | NA | NA | NA | 670 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET25 | NA | NA | 918 | 38 | 817 | 1168 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET6532 | NA | NA | NA | 1209 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT12 | NA | NA | 60 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 120 | 39 | NA |
| WT28 | NA | NA | 92 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 68 | 78 | NA |
| WT6 | NA | NA | 90 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1510 | 704 | 1057 |
| WT62 | NA | NA | 782 | 1245 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT6520 | NA | NA | 113 | 961 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT6536 | NA | NA | 488 | 396 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT6752 | NA | NA | 204 | 1009 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT7 | NA | NA | 774 | NA | NA | 222 | NA | NA | NA | NA | NA | NA | NA | NA | 14 | NA | NA |
| WT8 | NA | NA | 66 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 23 | 93 | NA |
And per sample per cell type (non-sphase):
| sample | LUMBAS | VFTISS | BAS | LUM | BL | LUM-B2 | AV | HS | BA | BAS-LEFT | BAS-RIGHT | LUM-LEFT | LUM-RIGHT | MIX | LUM-M | LUM-P | TISS |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ATMHET6182 | 60 | 700 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET17 | NA | NA | 573 | 18 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET43 | NA | NA | 808 | 1147 | 1285 | 984 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET49 | NA | NA | 1185 | 332 | 921 | 1138 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6139 | NA | 133 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6301 | NA | 171 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6389 | NA | NA | NA | NA | NA | NA | 469 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6410 | NA | NA | 329 | NA | NA | NA | 379 | 301 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6537 | NA | NA | NA | NA | NA | NA | 680 | 796 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6548 | NA | NA | NA | 1068 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B1HET6550 | NA | NA | NA | NA | NA | NA | 629 | 889 | 227 | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET16 | NA | NA | 236 | 845 | 752 | 723 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET18 | NA | NA | 1128 | 342 | 83 | 246 | NA | NA | NA | NA | NA | NA | NA | 16 | NA | NA | NA |
| B2HET21 | NA | NA | 1424 | NA | 1559 | 2 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET23 | NA | NA | 1027 | 688 | 1737 | 1211 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET232 | NA | NA | NA | 666 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET25 | NA | NA | 728 | 21 | 448 | 805 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| B2HET6532 | NA | NA | NA | 1208 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT12 | NA | NA | 44 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 93 | 33 | NA |
| WT28 | NA | NA | 85 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 41 | 75 | NA |
| WT6 | NA | NA | 48 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1245 | 510 | 822 |
| WT62 | NA | NA | 777 | 1233 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT6520 | NA | NA | 111 | 960 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT6536 | NA | NA | 488 | 396 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT6752 | NA | NA | 204 | 1009 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| WT7 | NA | NA | 616 | NA | NA | 220 | NA | NA | NA | NA | NA | NA | NA | NA | 9 | NA | NA |
| WT8 | NA | NA | 64 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 20 | 88 | NA |
I also plot some of this information in section 3.
2 QC
There appears to be a greater number of aneuploid cells in the unsorted WT6 sample.
Some of the cells look a bit noisy as shown below in the heatmaps and some individual cells. We’ll remove this sample from any further analysis for now.
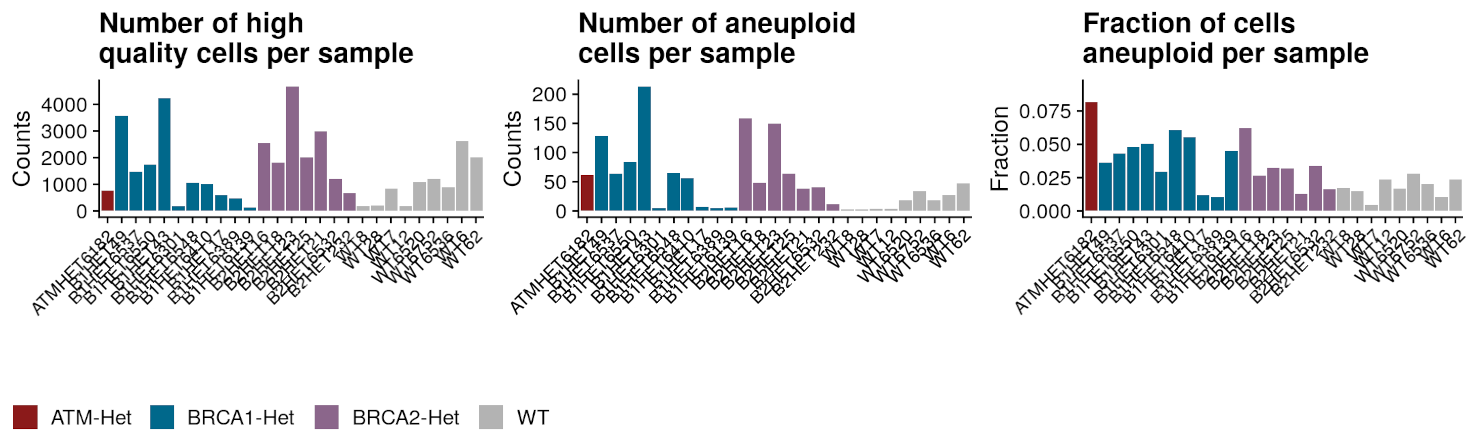
3 Summary
Here I’ll plot some overall summaries of the data including: number of cells per sample, fraction of cells aneuploid etc.
Total number of cells is 55116 and total number of non-sphase cells is 48175.
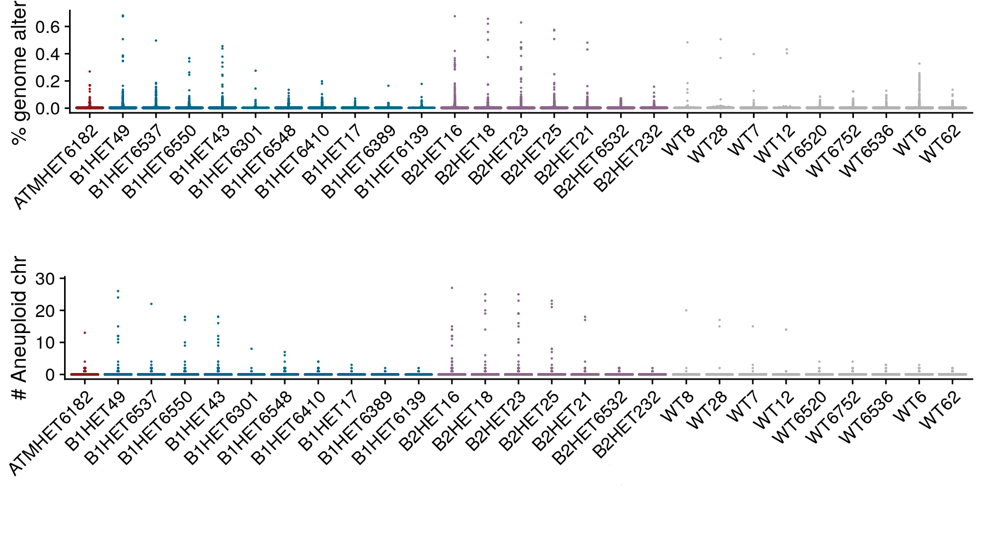
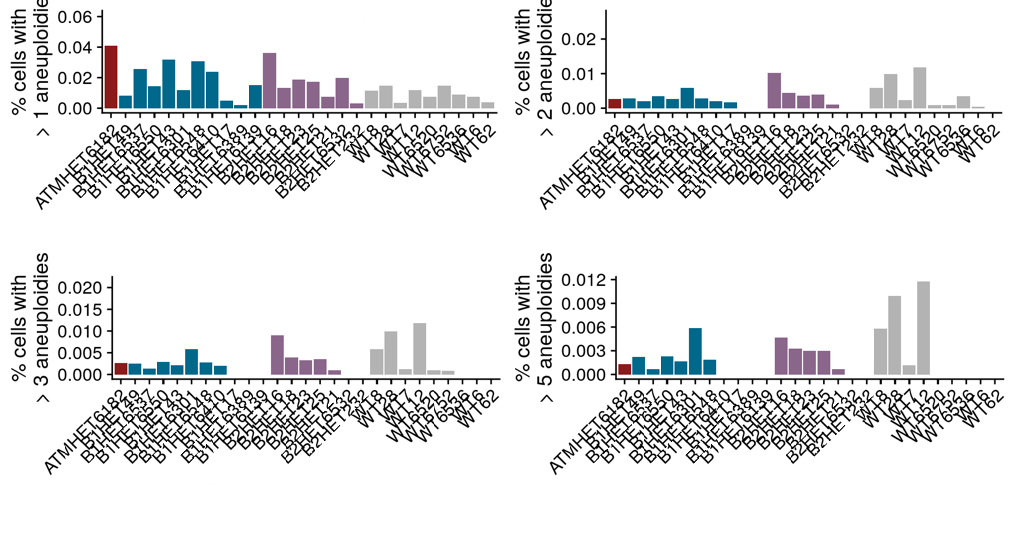
One of the most striking things about this data are the cells that have a large number of alterations. Here I’ll plot the fraction of cells with greater than X chromosomal changes, where X is 1,2,3,5.
Here are some of the abnormal WT cells.
WT12:
WT28:
WT8:
WT7:
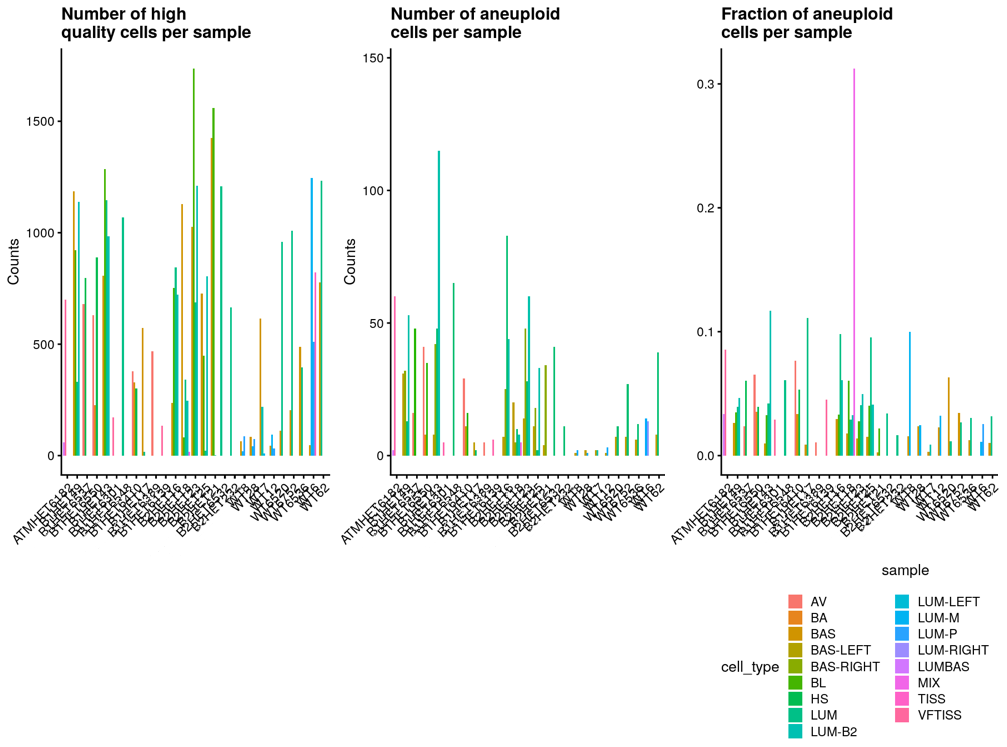
4 Summary per cell type
5 Copy number landscape
5.1 Full cohort
5.2 BRCA2 hets
5.3 BRCA1 hets
5.4 WT
5.5 Per patient
